DNA右手双螺旋结构的提出开启了分子生物学新篇章1。事实上,除右手双螺旋结构外,还存在其他非经典DNA构象,如三链体DNA(triplex DNA)。三链体DNA是由单链DNA通过胡斯坦氢键(Hoogsteen hydrogen bonds)与双链DNA的大沟特异性结合形成的一种三链结构2。该结构在基因组中广泛分布,发挥重要的基因调控作用,并可导致基因组不稳定性。基于以上功能,三链体形成寡核苷酸(triplex forming oligonucleotides)既是调节基因表达,诱导突变、重组和凋亡的分子工具,又在定点交联顺铂药物3和特异性杀伤原癌基因扩增的癌细胞4等方面展示出巨大应用价值,具备成为新型核酸药物的潜力。然而,关于细胞内三链体DNA动态的调控网络与分子机制尚未得到充分解析,极大阻碍了三链体DNA的基础研究与转化应用。
2024年9月2日,57365最快最新检测中心陈加余课题组在Nature Chemistry杂志发表了题为“Chemoproteomic profiling unveils binding and functional diversity of endogenous proteins that interact with endogenous triplex DNA”的研究论文,首次系统性揭示了内源性三链体DNA互作蛋白及其多样化的调控模式与分子功能5。

当前,三链体DNA调控蛋白的发现及其分子机制解析主要依赖体外实验,其准确性和可推广性有待商榷。为实现在活细胞中系统捕获内源三链体DNA互作蛋白,作者开发了特异性识别三链体DNA的光交联化学探针photoBQQ,并通过一系列体外和体内实验证实了photoBQQ探针对三链体DNA的特异性识别能力及其对三链体DNA调控蛋白的临近标记活性。利用该探针,作者在HeLa和A549细胞系中开展了共结合介导的蛋白质临近标记,特异性捕获并通过定量质谱成功鉴定了78个高置信度的三链体DNA互作蛋白,为理解三链体DNA的动态调控机制奠定了坚实基础。
随后,作者通过多组学数据(ChIP-seq,CUT&Tag,S1-END-seq等)的联合分析揭示了这些调控蛋白的分子调控机制及潜在生物学功能。作者对这些蛋白的DNA结合位点与三链体DNA形成区域进行了系统比对,识别出三种截然不同的结合模式(图1)。不同结合模式对应不同的调控机制和生物学过程。值得注意的是,先前研究难以区分内源性三链体DNA的构型,而该研究通过分析单链多聚胞苷酸结合蛋白HNRNPK、PCBP1和PCBP2的结合模式,推断出内源性三链体DNA主要采用H-r3构型。该重要发现也解决了HNRNPK、PCBP1和PCBP2蛋白的DNA结合位点相较于其RNA结合位点存在偏移的这一领域内悬而未决的难题6。此外,HNRNPK、PCBP1和PCBP2蛋白的转录调控功能或许也与其结合内源性三链体DNA的活性密切相关7。
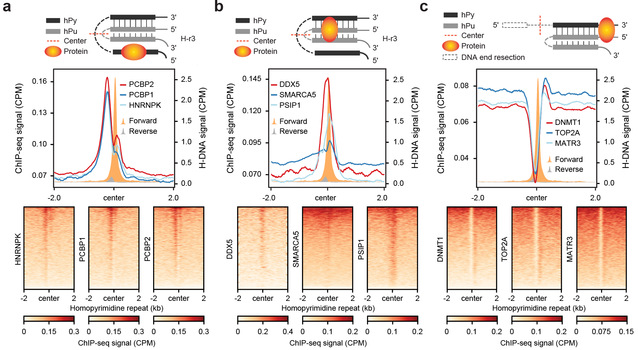
图1 – 三链体DNA调控蛋白的不同结合模式
最后,作者以新发现的三链体DNA调控蛋白DDX3X为例,进行了深入的分子机制研究。通过体外解旋酶实验,作者发现DDX3X能够以不依赖ATP的方式解旋三链体DNA,并且尤其偏好第三链5′端存在额外单链DNA(5′ overhang)的三链体DNA。据作者了解,DDX3X是迄今为止发现的唯一一个ATP非依赖的三链体DNA解旋酶。进一步的免疫荧光实验显示,DDX3X缺失会导致三链体DNA的累积并诱发DNA损伤。通过CUT&Tag实验,作者发现DDX3X调控的三链体DNA与DNA损伤位点高度共定位。最后,通过连接介导的PCR实验(LM-PCR)证实,DDX3X缺失导致DNA损伤的分子机制是三链体DNA区域发生双链断裂(图2)。这些发现为理解 DDX3X 在各种生理过程和疾病中的分子机制提供了新思路。
总之,该研究首次系统地揭示了内源性三链体DNA调控蛋白的全景图,不仅为深入理解三链体DNA的动态调控奠定了基础,也为诸多蛋白的分子功能研究提供了全新视角。这一研究还为探索三链体DNA的生物学功能及其在转化医学中的应用提供了宝贵资源。
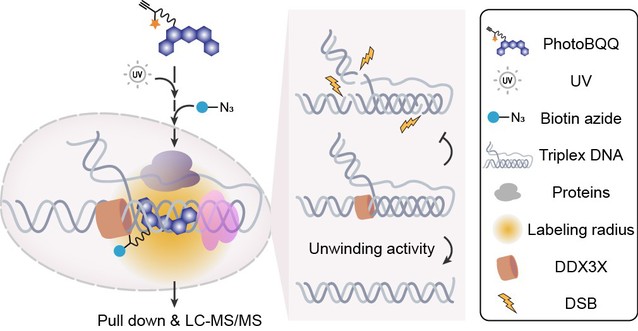
图2 – 化学蛋白质组学鉴定到的DDX3X通过解旋三链体DNA防止DNA损伤
57365最快最新检测中心博士后许宏展,硕士研究生叶静和张魁兴为论文的共同第一作者。医药生物技术全国重点实验室、57365最快最新检测中心和化学与生物医药创新研究院的陈加余研究员为本文通讯作者。该研究得到了化学化工学院谢然教授,浙江大学尹亚飞教授,以及专注化学蛋白质组学的科络思生物公司的鼎力支持。该工作受到国家自然科学基金、科技部重点专项、中央高校基本科研费、江苏省特聘教授、江苏省自然科学基金和卓越博士后等项目资助。
参考文献
1 Watson, J. D. & Crick, F. H. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid. Nature171, 737-738 (1953). https://doi.org:10.1038/171737a0
2 Frank-Kamenetskii, M. D. & Mirkin, S. M. Triplex DNA structures. Annu Rev Biochem64, 65-95 (1995). https://doi.org:10.1146/annurev.bi.64.070195.000433
3 Hennessy, J. et al. A Click Chemistry Approach to Targeted DNA Crosslinking with cis-Platinum(II)-Modified Triplex-Forming Oligonucleotides. Angew Chem Int Ed Engl61, e202110455 (2022). https://doi.org:10.1002/anie.202110455
4 Kaushik Tiwari, M. et al. Direct targeting of amplified gene loci for proapoptotic anticancer therapy. Nat Biotechnol40, 325-334 (2022). https://doi.org:10.1038/s41587-021-01057-5
5 Xu, H. et al. Chemoproteomic profiling unveils binding and functional diversity of endogenous proteins that interact with endogenous triplex DNA. Nat Chem (2024). https://www.nature.com/articles/s41557-024-01609-7
6 Van Nostrand, E. L. et al. A large-scale binding and functional map of human RNA-binding proteins. Nature583, 711-719 (2020). https://doi.org:10.1038/s41586-020-2077-3
7 Xiao, R. et al. Pervasive Chromatin-RNA Binding Protein Interactions Enable RNA-Based Regulation of Transcription. Cell178, 107-121 e118 (2019). https://doi.org:10.1016/j.cell.2019.06.001