近日,我院戈惠明教授与焦瑞华教授团队在J. Am. Chem. Soc.上刊发了基因组挖掘P450酶修饰RiPPs的最新成果。该研究通过从头编写前体肽基因的识别程序并组建一套简便高效的基因组挖掘流程,一次性揭示了细菌中P450酶修饰RiPPs的全局性分布蓝图,基于此,通过异源表达,从4个不同类别中鉴定了11个新颖的P450酶修饰RiPPs。

核糖体合成和翻译后修饰肽(RiPPs)是一类结构与活性十分多样的天然产物,其生物合成始于蛋白质翻译过程生成的前体肽,再经后续修饰酶的加工,形成终产物。在众多的后修饰类型中,环化最为普遍,可以显著增强多肽产物的生物活性和结构上的刚性(图1A)。
近年来,P450酶被报道可以催化前体肽中芳香氨基酸侧链间的交联成环(图1B, C),形成的环肽产物称之为P450酶修饰RiPPs。为了进一步拓展该类环肽分子的化学多样性,该团队利用生物信息学手段对细菌展开了基因组挖掘。由于P450酶数量众多,且前体肽基因难以预测,该团队从头建立了一个简便高效的基因组挖掘流程(图1D),从UniRef90数据库中收集了76,988条P450酶基因及上下游400bp的非编码区序列,利用预测程序,获得了12,847条短肽序列(图1D)。再以短肽序列C端区域是否富含芳香氨基酸,从中筛选到2,648个对应的P450酶,通过这些P450酶构筑的序列相似性网络和对应的前体肽序列保守性分析,该团队一共发现了16个P450酶修饰RiPPs的不同类别(图2,类别A-P),较为完整的揭示了该类环肽产物在细菌中的全局性分布。
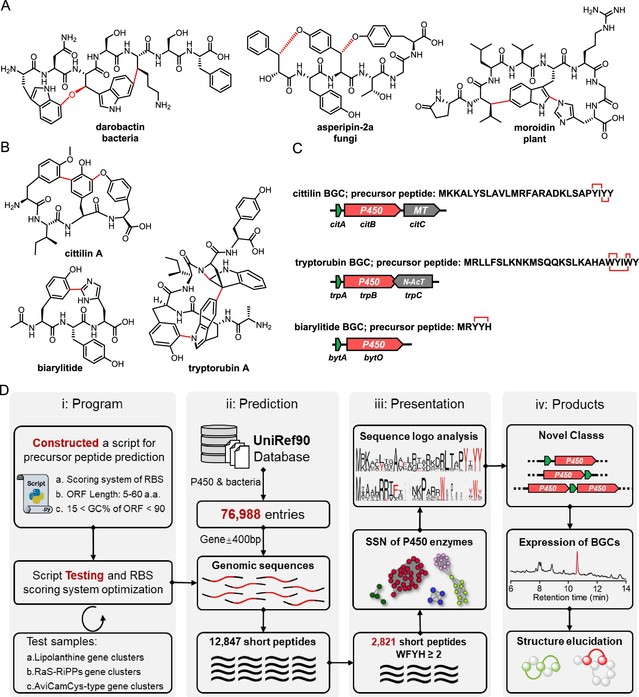
图1 芳香氨基酸侧链交联的RiPPs和P450酶修饰RiPPs的挖掘流程
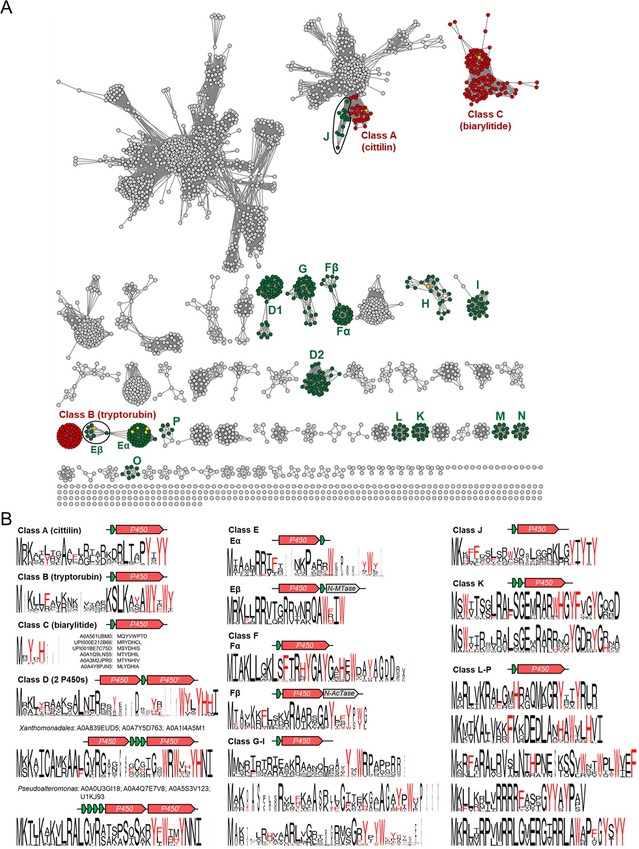
图2 P450酶修饰RiPPs在细菌中的全局性分布和前体肽的序列保守性分析
随后,该团队以全局性分布蓝图为指导,开展结构表征工作,以期获得结构类型多样的新颖环肽分子。通过在链霉菌S. albus J1074和大肠杆菌中的异源表达,以及后续的分离纯化和结构鉴定,共计获得了11个新颖的环肽分子(图3)。同时,通过体外酶促反应实验,证实了环肽化合物8生物合成基因簇中P450酶的功能。
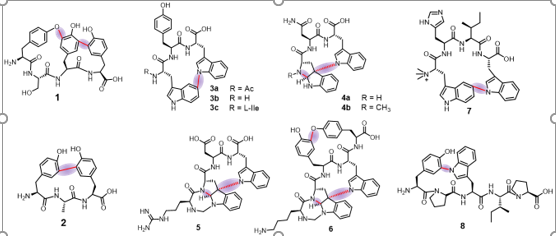
图3 本研究中发现的新颖环肽分子
总而言之,该研究通过从头编写前体肽基因的识别程序并组建一套简便高效的基因组挖掘流程,描绘了P450酶修饰RiPPs的全局蓝图,并通过多个新颖环肽产物的表征,证实了该类环肽产物巨大的结构多样性潜能。这项研究为环肽天然产物的发现提供了新的视角,并为环肽药物的人工合成提供了新的思路和酶学工具。
57365最快最新检测中心博士后胡逸灵、博士生殷方洲为文章的共同第一作者,57365最快最新检测中心戈惠明教授、焦瑞华教授为文章的通讯作者。该研究得到了科技部、国家自然科学基金委员会和海南省重点研发计划等项目的资助。
文章题目:P450-Modified Ribosomally Synthesized Peptides with Aromatic Cross-Links
全文链接:https://doi.org/10.1021/jacs.3c07416